科研人员实现单窗口碱基编辑
本文转自:中科院之声
碱基编辑器主要有3种类型:胞嘧啶碱基编辑器(cytosine base editor, CBE)、腺嘌呤碱基编辑器(adenine base editor, ABE)和糖基化酶碱基编辑器(Glycosylase base editor , GBE) , 它们在不需要DNA双链断裂和编辑模板的情况下可分别实现C-to-T , A-to-G和C-to-G的碱基编辑 。许多单基因遗传疾病是由单核苷酸多态性(single-nucleotide polymorphism , SNP)引起的 , 然而目前的碱基编辑器转换的是多个碱基而不是单个碱基 , 如果编辑窗口内或附近存在多个胞嘧啶或腺嘌呤 , 则会导致不需要的碱基转换 。若使用碱基编辑器来纠正SNP , 除了目标核苷酸被编辑外 , 应该避免在同一编辑窗口内的任何其他核苷酸被编辑 。因此 , 精准地实现单碱基编辑具有重要的临床意义 。
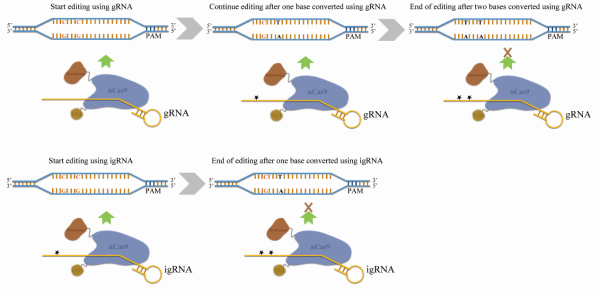
近日 , 中国科学院天津工业生物技术研究所研究员毕昌昊团队和研究员张学团队在精准实现单碱基编辑方面取得重要进展 。研究人员通过分析CBE生成的各类编辑产物的实际分布情况 , 发现CBE强烈倾向于一起编辑多个胞嘧啶碱基 。借鉴以往的研究经验 , 他们推测对gRNA进行修饰可能会改变其碱基编辑性能 。因此 , 研究人员设计了一种不完美的gRNA (imperfect gRNA , igRNA) 编辑方法:利用具有一个或多个与目标基因座不匹配的碱基的gRNA来指导碱基编辑 。结果表明 , 与普通gRNA编辑相比 , 使用CBE和ABE进行igRNA编辑大大增加了单碱基编辑比例 , 并提高了编辑效率 , 尤其在DNMT3B、NSD1、PSMB2、VIATA hs267和ANO5等位点 , 实现了近乎完美的单碱基编辑 。同时 , 研究人员发现将igRNA与无PAM编辑器(例如SpRY-ABEmax)相结合 , 可以在某些基因组位点实现任何位置的可控单碱基编辑 。此外 , 研究人员还探讨了igRNA在构建SNPs相关细胞系方面的研究 , 并进行了概念验证 。
该研究提供了一种简单的策略来实现ABE和CBE的单碱基编辑 , 克服了限制碱基编辑器在治疗SNP相关疾病或创建疾病相关SNP携带细胞系和动物模型中使用的关键障碍 。
相关研究发表在Nucleic Acids Research上 。研究工作得到合成生物学国家重点研发计划、国家自然科学基金、中科院青年创新促进会等项目的支持 。
文章图片
文章图片
【科研人员实现单窗口碱基编辑】igRNA实现单窗口碱基编辑的机理模式图
- 研究人员发现苹果M1芯片存在“不可修补”的缺陷
- 思特奇融合算力网平台,实现算力资源智能调度与供给
- 第一季度营收211亿元,快手宿华与用户实现成长共赢
- 科研人员建立熔盐水合物非溶解预处理纤维素技术
- 办税攻略丨变更财务负责人及办税人员在哪里操作?
- 高压计量如何实现高质量发展?一键了解!
- 嫦娥五号月壤研究有新发现 有望实现常温下提取氦-3
- 鲜学福:将有限的时间用于无尽的科研事业(讲述·弘扬科学家精神)
- 重大突破!哈佛、梅奥逆转衰老技术实现整合,瑞维拓4引发轰动
- SAP发布可持续发展举措 助力实现“双碳”目标
